Research Compendium(解析のパッケージ化)について
この記事は,Open and Reproducible Science Advent Calendar 2019の6日目の記事です。
研究で使用したデータや解析コードが公開されることが今後増えていくことかと思いますが,研究者が解析したそのままで公開すると第三者にはわかりにくいものになるかもしれません。データや解析コードの説明を含めたある程度決まった形式で解析をパッケージ化して配布することが必要になるかと思います。
困ったデータと解析コードの共有の例
具体例を出すと以下のようなフォルダを共有されても困るわけです。

まず,解析に関する説明文書が5つあります(「解析v2.txt」から「重要メモ.doc」です)。どれが見るべきファイルから判別できません。このフォルダを渡されたら,すべての説明文書を開いて解読するところから始める必要があります。
次に,データが4つあります(「データv1.csv」から「データ修正版v3.csv」までです)。異なるルールで記載された最終版があり,修正版には最も新しそうなバージョン番号がついています。そのため,どれが一番最新のデータなのか謎です。データの中身と解析コードを突き合わせていかないと判別できません。
最後に,解析コードのファイルが5つあります(「准教授Kを黙らせる解析.m」から「メインの結果_fin.R」までです)。「准教授Kを黙らせる解析.m」という心意気は良いとは思いますし,「分析with田中センパイ.m」からは田中先輩の人柄が伝わってきますが,研究の解析コードの名前としては情報がありません。また,「2番目に実施する.R」とありますが,じゃあ,最初にはどれを実施するの?と疑問がでてきます。多くの場合,解析コードは決められた順番で実施する必要があります(統計解析→前処理→データの読み込みという順番では正常に動作しないはずです)。その順番の情報がこれらのファイル名からはわかりません。
Research Compendium(解析のパッケージ化)
データと解析コードを整理する必要性は,多くの方にご納得いただけるかと思います。ただ,どのように整理するかというルールはないので,各研究者がそれぞれの美学に基づいて整理している感じかと思います。それがすごくわかりやすいものだったらいいのですが,癖があったりすると読みにくくなります(そして,その癖は自分では気が付かないかなと思います)。この形式が研究者によって異なると,読む側としてはちょっと認知的負荷が高いように思います。
そういうこともあり,再現性の高いデータと解析コードの共有に関して,Research Compendiumという考え方がでてきています。Research Compendiumは,以下の3つの特徴をもったデータと解析コードの整理法です。
- ファイルを研究者コミュニティにおいて広く流布された方法でまとめる
- データ,方法,出力を明確に分けた上で,それらの関係について明示する
- 解析が実施された解析環境を明確にする
Research Compendium(解析のパッケージ化)のやり方
Research Compendiumに基づいて,Marwick, Boettiger, & Mullen (2018)の"Packaging Data Analytical Work Reproducibly Using R (and Friends)"*1では,Rのパッケージ開発で使われている方法をデータと解析コードの共有方法として採用することを提案しています。Research Compendiumの1つ目である「ファイルを研究者コミュニティにおいて広く流布された方法でまとめる」は,Rのパッケージ開発の方法である必要はありません。しかし,Rパッケージ開発では,他の開発者とも共同作業ができるように,形式が決められており,そのファイル配置も合理的なものになっています。なお,ファイルの配置くらい自分で決めたいという気持ちもあるかもしれないですが,ここは個性を発揮しなくてもいいじゃないかなと思います。むしろ,すでに配置を決めたテンプレートがあると,あとは中身をいれるだけなので,簡単といえます。このように負担が小さくなるだけでなく,公開されたデータをその後のメタ分析などで活用する場合に,データと解析コードが同じファイル構造をもっている方が楽になると思います。
Marwick, Boettiger, & Mullen (2018)を参考にして,フォルダを作ると以下のようになります。公開するフォルダは,以下のファイルやサブフォルダ(サブフォルダには,/がついています)からなります。
- README 第三者がプロジェクトの全体像を理解できるように説明をするファイルです。どのdataに対して,どのfunctionを使って,どういうanalysisをしているのかを書きます。
- DESCRIPTION プロジェクトのメタデータや依存関係を記載するファイルです。
- LICENCE 公開したファイルは誰がどのように使えるものなのか説明するファイルです(MITライセンスなどよく使われるものを参考にするとよいです)。
- data/ 使用したデータを配置するフォルダです。可能であれば,最も前処理をしてないデータ(生データか匿名化処理後のデータ)だけを配置して,前処理後のデータは置かない(その処理はanalysisで行う)。
- analysis/ 使用した解析コードを配置するフォルダです。解析コードが複数ある場合は,どういう順番で実行するのか分かるようなファイル名にしておくとよいです。
- function/ プロジェクトで使用した関数を配置するフォルダです。 Marwick, Boettiger, & Mullen (2018)の場合は,R/となっていますが,PythonやMatlabも使うこともあるので,functionにしています。

上記のようにデータや解析コードを準備すれば,他の研究者は,まずREADMEを読んで,次に,analysisフォルダを開いて,analysis01.Rmdから順番に実行すれば良いです。その後,functionが気になったりしたら,そっちを確認することもできます。なお,上記のResearch Compendiumは,Marwick, Boettiger, & Mullen (2018)の例の中でも最小限のものになるので,より大規模なものなどを参考にしたい場合は,Research Compendiumで探すと良いかと思います。日本語の例としては,著者の研究室で取り組んだ追試研究のデータと解析コードの共有リポジトリ(https://github.com/ykunisato/somatori_kunisato_2019_replication_study)がありますので,参照ください。
今回解説した解析のパッケージ化は,再現可能な心理学研究入門でも触れています。データ解析の再現可能性の全体像を把握したうえで,上記の解析のパッケージ化の位置づけを知りたい場合は,読んでいただけたら幸いです。
Enjoy!
*1:Marwick, B., Boettiger, C., & Mullen, L. (2018). Packaging Data Analytical Work Reproducibly Using R (and Friends). The American Statistician, 72(1), 80–88.
共有を前提としてデータを管理する
この記事は,Open and Reproducible Science Advent Calendar 2019の5日目の記事です。
データ解析の再現可能性を高めるためには,生データから最終的に論文で報告されている結果までの処理過程を再現可能にした上で公開する必要があります。今回は,その方法について説明します。
データの管理については,いろいろな方法があるとは思いますが,Wilson et al., (2017)*1は,データ管理の推奨実践として以下を挙げています。
- 生データを保存する
- 生データは1つ以上の場所にバックアップをとっておく
- 第三者に提供しやすい形式にデータを整える
- データを解析しやすいように整える
- データ処理に用いたすべてのステップを記録する
- 複数のデータセットを使用することを想定して,ユニークな識別子を使用する
- 他の研究者がアクセスし引用できるようにデジタルオブジェクト識別子(Digital Object Identifier: DOI)の付与ができるリポジトリにデータを登録する
以下では,Wilson et al., (2017)の推奨実践について詳しく説明します。
1.生データを保存する
2.生データは1つ以上の場所にバックアップをとっておく
可能な限り生データを公開することが重要です(もちろん,個人情報が適切に保護できているのであればの話です)。色々なデータの整形を行ったきれいなデータを公開したくなりますが,その「色々なデータの整形」にミスが入り込むことが多いです。もし,いろいろなデータ整形に致命的なミスがあるものの整形後のデータを公開しているとその致命的なミスが分からなくなってしまう可能性があります。そこで,まずは,なんらかの処理をしたデータを保存するのではなく,大元の生データをしっかり保存しましょう。また,生データは1つ以上の場所にバックアップをとっておきましょう。電子データはどのように保存してもちょっとしたことでデータが破損する可能性があります。必ず,複数箇所にデータを保存するようにしておくと安心です。
3.第三者に提供しやすい形式にデータを整える
4.データを解析しやすいように整える
生データそのままでは統計解析可能な形式になってないかもしれません。そこで,第三者にも理解しやすい形式に整形します。変数名やファイル名もわかりやすい名前にします。データ整形にあたっては,データサイエンティストのHadley Wickhamが提唱する整然データ(Wickham & Grolemund, 2016)を意識して整形を行うと良いです。整然データとは,1つのセルには個々の値が入り,1つの列には個々の変数が入り,1つの行には個々の観測が入るという原則の元で整えられたデータのことを指します。整然データになっていれば,人間も読みやすいですし,機械も処理しやすい形式になりますので,統計解析に利用しやすいです。
5.データ処理に用いたすべてのステップを記録する
6.複数のデータセットを使用することを想定して,ユニークな識別子を使用する
データの整形過程から統計処理までのすべての処理過程を記録します。この処理過程がすべて残せるかどうかが再現可能性においては重要です。特定の商品名は避けますが,よく使用されるGUIベースの統計ソフトの場合(パソコンの画面でポチポチクリックする系のソフト),なんかの方法で別途記録を残しておかないと,すべての処理過程が残りませんので,再現不可能になります。その点,RなどのCUIベースのソフトは(コマンドを打ち込んで操作する系のソフト),すべての処理過程を明示的に残せるので再現可能性においては優れています。さらに,Rの場合はR Markdownを用いると,データに対して行った操作だけでなく,なぜそれを行ったかなどの説明も残すことができ,さらに再現可能性を高められます。R Markdownについては,高橋康介先生の「再現可能性のすゝめ」を薦めます。なお,ある個人のデータが複数のデータセットに分かれている場合があります。例えば,質問紙のデータベース,個人属性情報のデータベース,MRIのデータベースなどが別々にあることもあります。この場合に,それぞれが特定の人のデータであると識別できるように,ユニークな識別子を使います。これによって,各データベースを連結することができます。
7.他の研究者がアクセスし引用できるようにデジタルオブジェクト識別子(Digital Object Identifier: DOI)の付与ができるリポジトリにデータを登録する
データを公開する先は個人のHPなどではなく,他の研究者がアクセスしやすく引用しやすい比較的永続性のあるリポジトリを使うことをおすすめします。例えば,figshare (https://figshare.com/)やZenodo (https://zenodo.org/)などがあります。figshareもZenodoもDOIが付与されますので,恒久的に引用することができるようになります。このようにしておくと,後の研究者がデータの再利用する時に引用されるようになるので,自身の研究のインパクトが増します。
今回解説したデータの管理方法は,再現可能な心理学研究入門でも触れています。データ解析の再現可能性の全体像を把握したうえで,上記のデータ管理の位置づけを知りたい場合は,読んでいただけたら幸いです。
Enjoy!
*1:Wilson, G., Bryan, J., Cranston, K., Kitzes, J., Nederbragt, L., & Teal, T. K. (2017). Good enough practices in scientific computing. PLoS Computational Biology, 13(6), e1005510.
心理学黙示録の四騎士
この記事は,Open and Reproducible Science Advent Calendar 2019の4日目の記事です。
ヨハネの黙示録では四騎士は,疫病,戦争,飢饉,死ですが・・・*1
Dan Quintanaによると,心理学黙示録では,再現可能性,一般化可能性,理論,測定だそうです。再現性をなんとかすれば終わりというわけではなさそうですね。
The four horsemen of the psychological science apocalypse pic.twitter.com/3I2XrgQWt3
— Dan Quintana (@dsquintana) 2019年11月22日
このツイートを見た時に,ちょうどDaniel Lakensの数年前のブログ記事をみてて,ほー,今後もこんなにクライシスが!!!((( ;゚Д゚)))ブルブル,と思っていたところでした。
Dan Quintanaが紹介している論文のリンクを以下につけておきます*2。
- 再現可能性
A manifesto for reproducible science | Nature Human Behaviour
- 測定
https://www.psychologicalscience.org/observer/measurement-matters
Questionable Measurement Practices (QMPs) https://psyarxiv.com/hs7wm/
Questionable MAnipulation Practices (QMAPs) https://psyarxiv.com/t7ev9/
- 一般化可能性
- 理論
RstanにDockerはいいゾ
この記事は,Stan Advent Calendar 20192日目の記事です。
昨年もDockerで記事を書いたのですが,実は思ったよりDockerは活用されてないぞと思うことが何度かありました。そこで,今年もDockerについて書きます。
今年は,MacOSのCatalinaがリリースされました。Catalinaでは色々と新機能が追加されたので,ワクワクとした気持ちで試したMacユーザーも多かったのではないでしょうか?しかし,CatalinaにアップグレードするとStanが動作しないという問題があり,軽い気持ちでアップグレードしたStanユーザーを絶望の淵に追い込みました(Stan Advent Calendar 2019の1日目のkosugittiさんの記事でその辺の経緯がまとめられています)。こういう時こそ,以下のStan公式のツイートにあるように,Dockerなどの仮想コンテナを使っていると安心です。
Mac users: upgrading to Catalina can break Stan 😟🔥 We strongly advise against updating. If you already updated, follow the thread at https://t.co/ST6OAD40r6 for possible workarounds 🧯(so far only working through Docker seems to consistently help). Sorry for the inconvenience.
— Stan (@mcmc_stan) 2019年10月23日
ただ,Dockerがどのように便利か説明しても,Dockerの仕組みを説明しても,あんまり活用されない気がします。おそらく,新しい技術の導入かかる時間的・認知的コストを考えると,現状で困ってないので,Docker使わなくてもいいかなという判断になるのではないかなと思います。今,少しギクッとした方は,ぜひとももう少し読んでください。
以下に紹介するようにやれば,MacユーザーがDockerのデスクトップアプリをインストールするところから,StanでEight Schoolsを実施するまでに10分もかかりません*1。試して失敗したところで,MacOSのアップデートのような他のソフトへに影響もありません。Twitterで無駄に浪費する10分を私にいただいて,試してみませんか?
さあスタートです(ストップウォッチスタート)。docker hubにアクセスしてください!
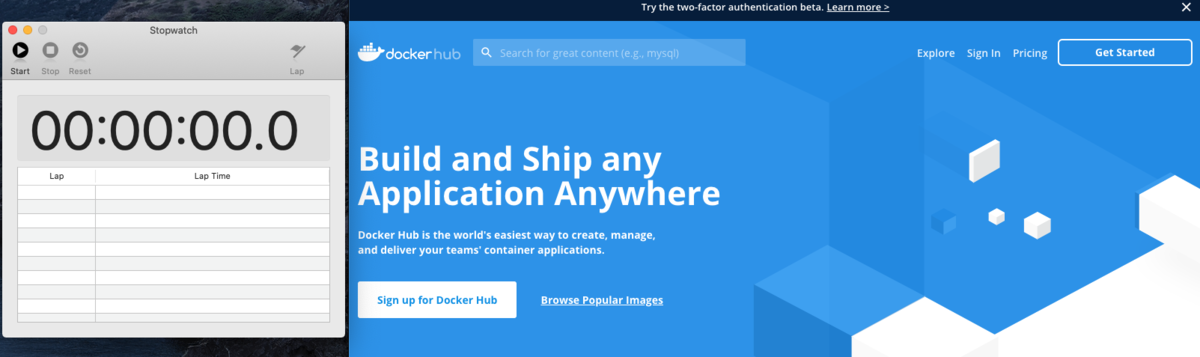
Sing up for Docker Hubをクリックします!そして,必要事項を入力します(IDとかメールとかパスワードとかです)。
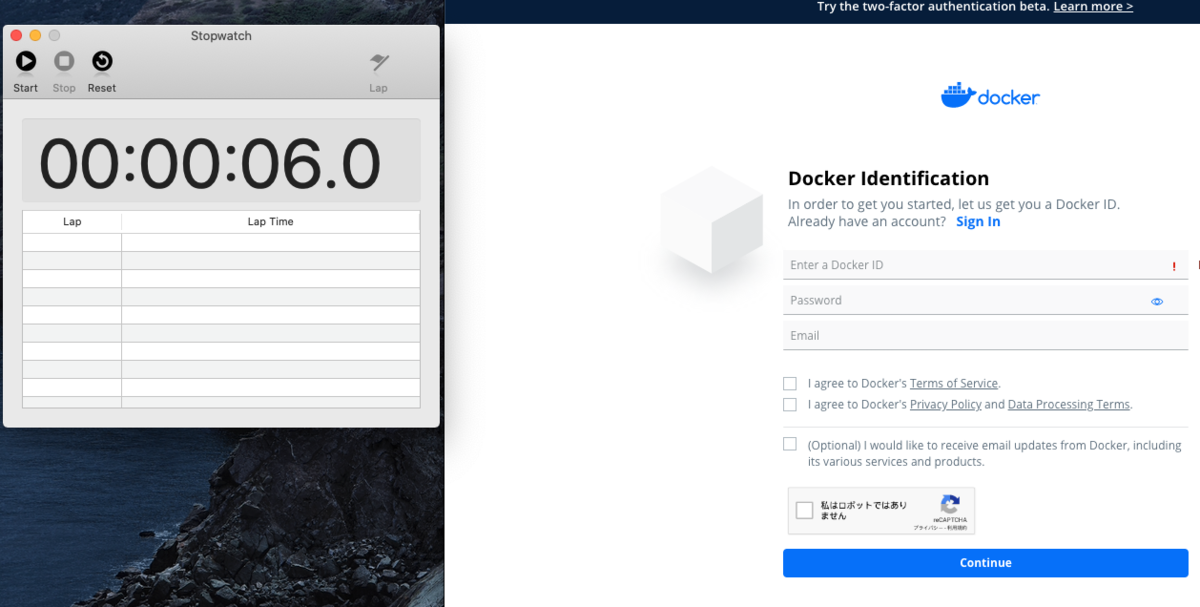
登録したらメールとか届くので確認します。Docker hubにログインします。
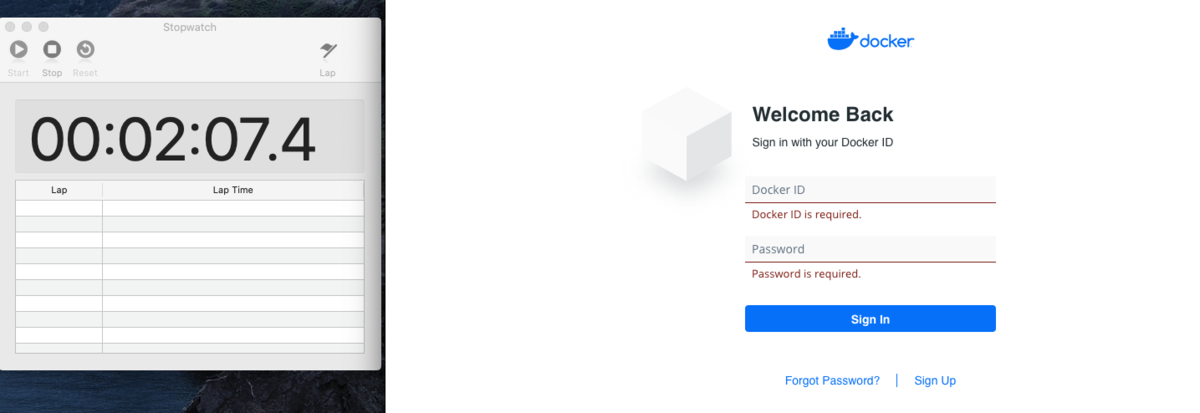
ログインしたら,どこかに「Download Docker Desktop」があるので探してクリック,以下のようなページに移動して,「Download Docker Desktop for Mac」が出てくるのでクリックします。Docker Desktopのダウンロードが始まります。

ダウンロードしたものをダブルクリックしたりして,以下のような状態までもっていって,アプリケーションフォルダにコピーします。

アプリケーションフォルダのDocker Desktopアプリをダブルクリックします。

Macの右上のメニューバーでDockerがもぞもぞ動いています(開始しています)。落ち着いたら,クリックしてみると,Sing inってのがあるので,そこをクリックして,先程作ったDocker hubのアカウントを追加します。
ここまでDocker Desktopの準備で5分ほどかかりました。とはいえ,普通のアプリのインストールと変わらないかと思います。
続いて,ターミナルを起動します。なんか黒い画面が出てきて「怖いな,怖いなあ」と思うかもしれませんが,1行コードを打つだけなので我慢してください。

今回は,Rstanやその他色々とパッケージの入ったykunisato/paper-r-jpを使います(私が準備したDocker コンテナです)。これ1つあれば,大体のことはできます。その他にもコンテナは用意しているので,適宜好きなものを使用ください。一応解説は,ykunisato/paper-r-jpにも書いてありますが,以下をターミナルに打ち込むだけです(パスワードとコンテナ名は好きなものを使ってください)。
docker run -e PASSWORD=パスワード -p 8787:8787 -v $PWD:/home/rstudio -d --name コンテナ名 ykunisato/paper-r-jp
例えば,以下のようにパスワードとコンテナ名を変更します(パスワードをpass,コンテナ名をpaper)。なお,パスワードはちゃんとしたのを設定ください。
docker run -e PASSWORD=pass -p 8787:8787 -v $PWD:/home/rstudio -d --name paper ykunisato/paper-r-jp
しばらく待ちます。まあまあ重いファイルをダウンロードするので,すごい遅い回線だと時間かかります(それで10分超えちゃうかも・・・)。以下のような感じになったら終了です。これですでにRstanの入ったRstudio serverのDockerコンテナが走っています。

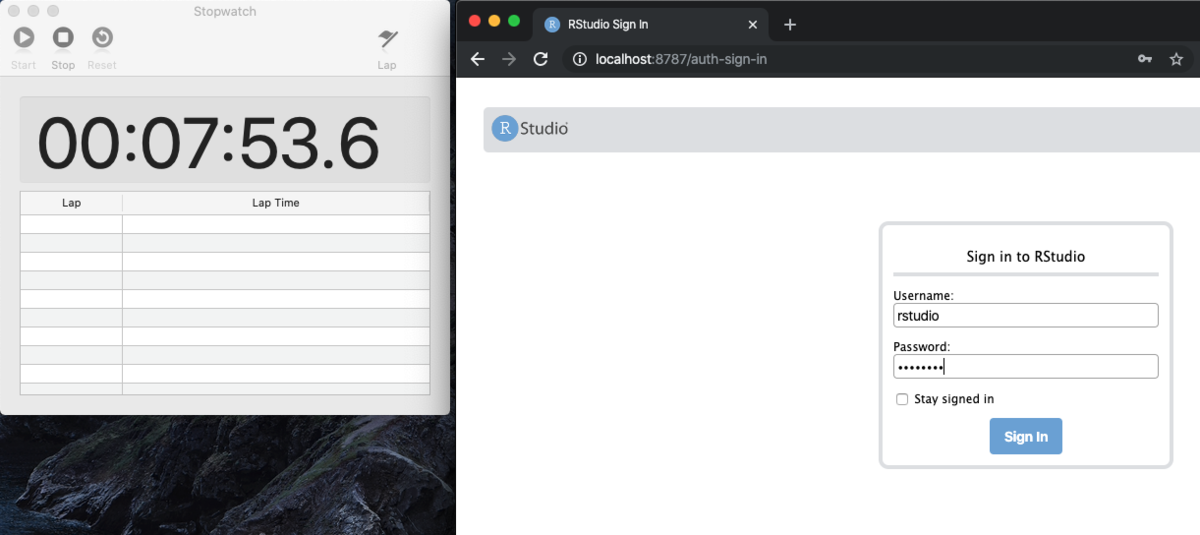
ご自身のインターネットブラウザ(Chromeとか)を開いて,http://localhost:8787/ と打ち込んでください。以下のようなRstudioのログイン画面が出てくるので,Usernameに"rstudio",Passwordに上記で設定したパスワードを入れてください。
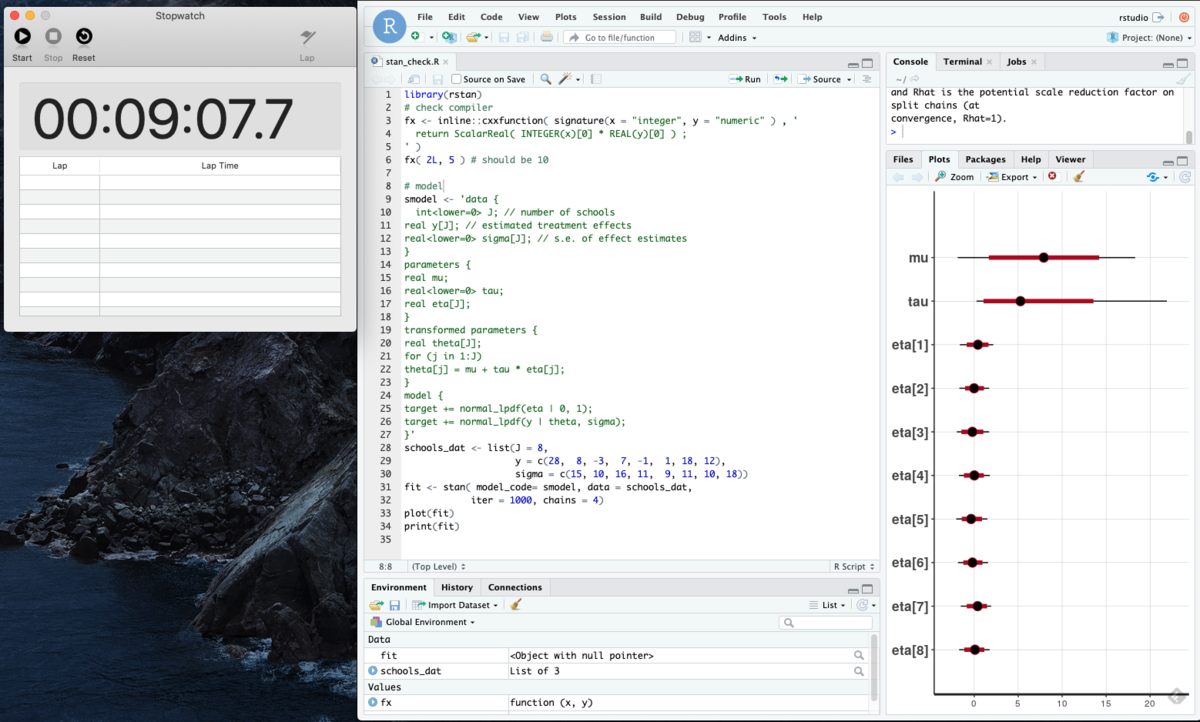
さあ,ちゃんとRstanが動くか調べてみましょう!RStan Getting Startedで使われるEight Schoolsが動作するか以下をConsoleにコピペしてみましょう。
library(rstan) # check compiler fx <- inline::cxxfunction( signature(x = "integer", y = "numeric" ) , ' return ScalarReal( INTEGER(x)[0] * REAL(y)[0] ) ; ' ) fx( 2L, 5 ) # should be 10 # model smodel <- 'data { int<lower=0> J; // number of schools real y[J]; // estimated treatment effects real<lower=0> sigma[J]; // s.e. of effect estimates } parameters { real mu; real<lower=0> tau; real eta[J]; } transformed parameters { real theta[J]; for (j in 1:J) theta[j] = mu + tau * eta[j]; } model { target += normal_lpdf(eta | 0, 1); target += normal_lpdf(y | theta, sigma); }' schools_dat <- list(J = 8, y = c(28, 8, -3, 7, -1, 1, 18, 12), sigma = c(15, 10, 16, 11, 9, 11, 10, 18)) fit <- stan( model_code= smodel, data = schools_dat, iter = 1000, chains = 4) plot(fit) print(fit)
終わりました!10分もかからず,9分程度で,Dockerの準備からRstanの実行までいけました!簡単でしょう?
継続的にDockerを使う場合は,あと何個かDockerのコードを覚える必要がありますが(コンテナの開始・中止・削除など),それはまた今度にします。
10分以内でのRstan環境の構築,お試しいただいてはいかがでしょうか?
Enjoy!
JCORSはじめました!!!
この記事は,Open and Reproducible Science Advent Calendar 2019の1日目の記事です。
2019年11月11日から,日本でオープンサイエンスや再現可能性に関してオンラインで情報共有をするJapanese Community for Open and Reproducible Science (JCORS)をはじめました。
JCORSは,人を対象とした研究を行う研究者・実務家が透明性の高い研究を行う上での情報共有をする研究コミュニティです。基本的にはSlack上で情報共有をして,そこで共有・議論された情報をOSFのWikiで順次公開していく予定です。
まだ,準備中の部分も多いですが,以下のOSFのWikiページには,オープンサイエンスや再現可能性に関する情報がまとめてありますので,最初に学ぶ際に役に立つのではないかと期待しています。興味を持たれた方は,是非とも以下のOSFのWikiページにあるメールフォームからご参加ください!
JCORSのTwitterアカウントもあるので,よろしければ,フォローください。
少しだけ会の立ち上げとその後の経緯を記載しておきます。私(国里愛彦)は,2019年度は在外研究制度を活用して,アムステルダム大学心理学科Psychological Methodsに滞在しています。滞在中に,Open Science Community AmsterdamのイベントやSociety for the Improvement of Psychological Science (SIPS)に参加して,日本の心理学においてもオープンサイエンスのコミュニティが必要であることを感じました。
帰国してから始めても良かったのですが,帰国したら大学のお仕事がすごーく忙しくなりそうな雰囲気を察知したので,思いついたタイミングで始めてみました。当初は,Open Science Community Amsterdamを参考に,Open Science Psychology Community in Tokyo (OSPCT)という名前で始めました。JapanにするかTokyoにするか,さらには日本の勤務先のKanagawaとかKawasakiにするか悩んだのですが,Japanを背負うのはちょっと気が引けました。もし会を初めたものの会員にが自分だけだった場合を考えると,自意識がJapanを選ぶことを邪魔しました。そして,Kanagawaだと少しローカル感がでちゃうので,Tokyoにしました。今思うとTokyoを背負うのも荷が重い気がしますが,その時はそれに気がつかず,衝動的にOSFのアカウントを作成&ツイートをしていました(要するに,そこまで考えられた名前ではないです)。
心理学の研究者や心理士が透明性の高い研究手法の情報を共有するためのオンラインプラットフォームとして,Open Science Psychology Community in Tokyoってのを作ってみました。https://t.co/hcecnjhsnk
— Mr. CCP (@akabuchiyk) 2019年11月11日
Wikiの共同編集者を募集しています。slackも作ったので,メールフォームから申請ください〜
始めてみたところ,思ったよりも多くの方にご参加いただけたのと(すでにオープンサイエンスや再現可能性に詳しい方の参加もあり,心強かったです),思ったよりも様々な分野の方にご参加いただけました。さらに,オンラインのコミュニティということで東京にお住まいではない方も多かったです(私も2019年度は東京に住んでいませんし・・・)。そうなっていくると東京に限定するのも心理学に限定するのも微妙に思えてきて,Open Science Psychology Community in Tokyo という名前への違和感が強くなってきました。
そこで,新しい会の名前を募集し,その上で投票をすることにしました(投票の経過などはJCORSのslackに参加すると見れます。ちょっとグダグダしたところもオープンになっています)。そして,11月15日には投票の結果,Japanese Community for Open and Reproducible Science (JCORS)に決まりました。ということで,以下のツイートにあるように,5日目にして改名ということになりました。
開始5日目にして, Japanese Community for Open and Reproducible Science (JCORS)に改名することになりました (*゚ー゚)> https://t.co/hcecnjhsnk
— Mr. CCP (@akabuchiyk) 2019年11月15日
東京や心理学に限定せず,データに基づいた研究や実践をされていて,オープンサイエンスや再現性に関心のある方にご参加いただけましたら幸いです https://t.co/xprl3hS3TW
心機一転,Japanese Community for Open and Reproducible Science (JCORS)として再出発することになり,会のステッカーも決めました。オープンサイエンス(オープンソース)系で流行っている,Hexagon stickerを作成しました。安易ですが,日本ということで富士山をデザインしました。なお,富士山はkohskeさんのブログを参考にggplot2で描いて(雲は2変量正規分布を使いました),ステッカーはhexStickerパッケージを使いました。

OSFにステッカーのRコードを共有しているので,興味がある方はダウンロードして遊んでみてください(このRコードを使えばステッカー作成が再現されます)。
最後に,JCORSでは,アドベントカレンダーを行っています。このあとも,順次オープンサイエンス関係の記事がアップされていきますので,クリスマスまで楽しみになさっていてください。
Enjoy!
DockerでInkscape
図の作成にあたってはInkscapeを使うのですが,macOSのMojaveにアップグレードしてから,うまくインストールor起動ができなくなりました。ここで頑張るのも面倒なので,InkscapeもDockerで運用したいと思って少し調べてみました。
すでに,InkscapeのDockerファイルが準備されていたので,以下のDockerファイル(johnodon/inkscape)を使います。
johnodon/inkscapeは,hurricane/dockergui上にInkscapeをいれたものです。hurricane/dockerguiは,以下にあるように,ウェブブラウザ上でGUIアプリケーションを動かすためのDockerファイルになります。
This Docker image makes it possible to use any X application on a headless server through a modern web browser such as chrome.
さて,さっそくターミナルに以下を打ち込んで,pullします。
docker pull johnodon/inkscape
pullが終わったら,以下を打ち込んで,runします。
docker run -d -p 8080:8080 -p 3389:3389 -v ~:/home -e "TZ=Asia/Tokyo" --name inkscape johnodon/inkscape
そして,ウェブブラウザ上のURLバーに以下を打ち込みます。
localhost:8080
以下のような画面が開いて,Inkscapeを選択して,「OK」を押します。
そうすると,以下のようにInkscapeが起動します。
なお,run時に-v ~:/homeと指定しているので,homeに本体Macのホームが追加されているので,ファイルのやり取りも簡単です。
ただし,すでにあるファイルを読み込んでみたら日本語が表示されてない...日本語環境にしないといけないみたい... 英語の図や文字を入れない場合は問題ないですが,日本語を入れる場合はもう少し工夫が必要そう(また,Dockerファイルを自作するかな・・・)。 この辺は,また後日に。